I- Les patients :
Vingt et un patients atteints de N-IgA primitive ont fait l’objet de cette étude. Il s’agit de 6 femmes et 15 hommes, d’âge moyen 35 ans avec des extrêmes allant de 18 à 60 ans.
La durée d’évolution de la maladie varie de 22 ans à 6 mois avec une moyenne de 5,54 ans. Parmi nos patients, 16 soit 76,19% patients présentaient, au moment de l’étude, une forme grave de la néphropathie avec une HTA et/ou insuffisance rénale initiale.
Le diagnostic de la N-IgA a été fait par l’examen en immunofluorescence des PBR, réalisées dans le Service de Néphrologie de l’hôpital Charles Nicolle, montrant des dépôts caractéristiques glomérulaires constitués majoritairement d’IgA.
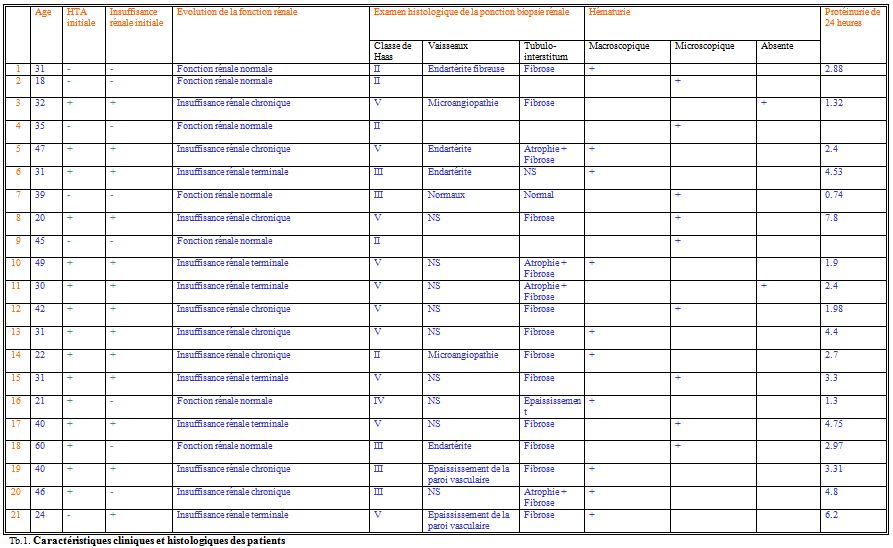
Les caractéristiques cliniques et histologiques de ces patients sont illustrées dans le tableau n°1.
Tb.1. Caractéristiques cliniques et histologiques des patients
L’examen des PBR en microscopie optique a révélé plusieurs types de lésions, permettant de classer les malades selon la classification de Haas (5), comme suit : 5 cas classe II, 5 cas classe III, 1 cas classe IV et 10/21 (47,62%) patients étaient de classe V histologique (Tb.1).
II- Les témoins :
Le groupe des témoins était constitué par des sujets normaux non apparentés, donneurs de sang :
-100 sujets pour les polymorphismes des gènes de l’IL-1, IL1-Ra et Apo-1/Fas.
-110 pour celui de l’exon 1(+49) du gène de la molécule CTLA-4.
III- Méthodes :
1. Séro-immunologiques :
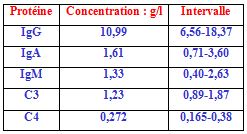
a- Dosage des immunoglobulines sériques : IgG, IgA, IgM et des fractions C3, C4 du complément par néphélométrie : MININEPH TM Kits (The Binding Site. Ltd.UK):
Principe :
Cette méthode utilise la diffraction d’un rayon Laser par les complexes immuns (protéine à doser-antisérum spécifique) formés en milieu liquide. La concentration de la protéine est proportionnelle à l’intensité de la lumière diffractée. Elle nécessite une courbe étalon tracée à partir de concentrations standard de la protéine à doser.
Résultats :
Les concentrations en protéines sont exprimées en g/l. Les valeurs normales (VN) sont consignées dans le tableau suivant :
b- Recherche des complexes immuns circulants (CIC) et leur typage : par la précipitation au polyéthylène glycol (PEG).
Principe :
Méthode physico-chimique qui tient compte de la taille et de la solubilité des CI.
Ces derniers sont représentés par des agrégats de haut poids moléculaire et peuvent par conséquent être séparés par précipitation avec le PEG. Le PEG est un polymère linéaire non chargé qui précipite, en solution, les protéines en fonction de leur taille et leur conformation moléculaire. La concentration en PEG utilisée est celle qui précipite les immunoglobulines (Ig) agrégées et qui maintient les globulines natives en solution. Le précipité de CI est isolé par centrifugation à froid.
L’analyse de la composition des CI en Ig et en protéines du complément est faite par la technique d’immuno-diffusion double type Ouchterlony utilisant des antisérums spécifiques aux protéines.
c – Recherche des auto-anticorps : anticorps antinucléaires (AAN), anticorps anti-mitochondries (AAM), anticorps anti-muscle lisse (AML) par immunofluorescence indirecte (IFI) sur coupes d’organes :
Principe :
Le sérum convenablement dilué, contenant éventuellement des auto-anticorps, est incubé sur des coupes d’organes portées à congélation. Les complexes antigènes- anticorps (Ag-Ac) sont révélés par des anti-IgG humaines marquées à la fluorescéïne.
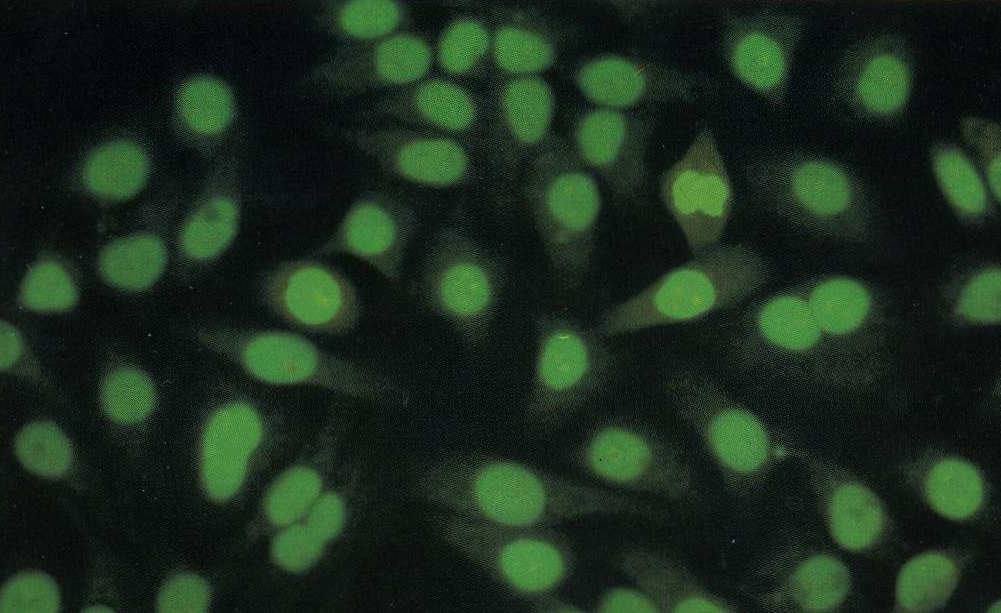
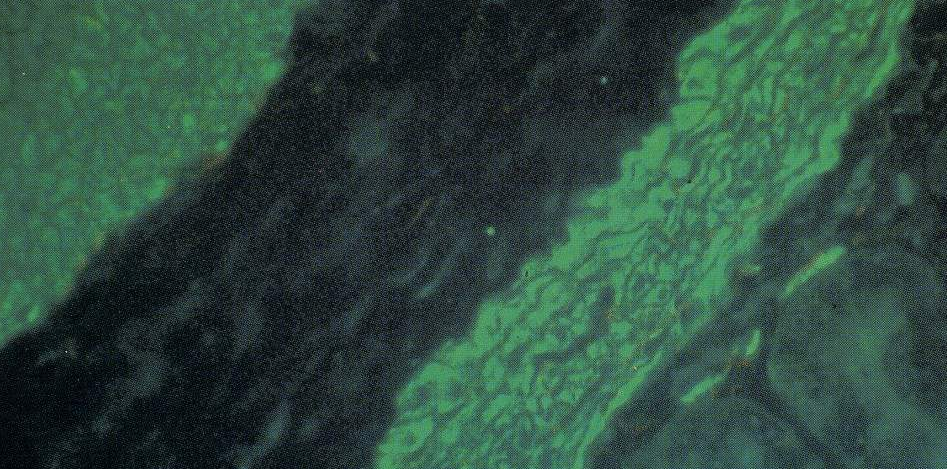
Les complexes sont identifiés grâce à leur localisation précise au niveau des structures du substrat. Ce dernier est constitué de foie de rat et/ou de cellules Hep2 pour les AAN, rein de rat pour les AAM et estomac de rat pour les AML (Fig 1 et 2).
Les cellules Hep2 provenant de culture d’un cancer trachéal humain, présentent par rapport aux coupes d’organes (surtout pour la détection des AAN) de nombreux avantages : cellules directement cultivées sur les lames de verre, population cellulaire homogène à grands noyaux de morphologie identique d’une cellule à une autre avec des nucléoles bien visibles, permettant la détection d’AAN réagissant avec des constituants cellulaires présents uniquement dans les cellules en division.
Fig.1 : AAN positifs sur des coupes de foie de rat.
Fig.2 : AML positifs sur des coupes d’estomac de rat.
d – Recherche des anticorps anti-cardiolipine de type IgG (aCL) et des facteurs rhumatoïdes (FR) de type IgM par méthode immuno-enzymatique type ELISA :
Principe :
Dans des microplaques sensibilisées par l’Ag (cardiolipine pour les aCL et IgG humaines agrégées pour les FR), les sérums des malades contenant éventuellement les Ac sont incubés. Les complexes sont révélés par des IgG (pour les aCL) et des IgM (pour les FR) humaines couplées à l’enzyme (la peroxydase dans notre cas).
L’activité enzymatique est révélée par le substrat (O.phénylènediamine ou TMB) donnant une coloration, en cas de réaction positive, mesurable au spectrophotomètre à 492 nm ou 450 nm respectivement.
Résultats :
La quantité de l’anticorps est dans ce cas proportionnelle à la densité optique de la coloration développée. Les résultats sont exprimés soit sous forme semi-quantitative, en nombre de croix, par rapport à un témoin positif standard soit sous forme quantitative en UI par rapport à des étalons.
2. Polymorphismes génétiques :
Dans notre étude plusieurs polymorphismes ont été analysés :
– Polymorphisme à la position -889 du gène de l’IL1α
– Polymorphisme à la position +3954 du gène de l’IL1β.
– Polymorphisme en VNTR au niveau de l’intron 2 du gène de l’IL 1-Ra.
– Polymorphisme à la position -670 du gène Apo1/Fas.
– Polymorphisme à la position +49 de l’exon 1 du gène CTLA-4.
a. Polymorphisme à la position (-889) du gène de l’IL1-α par PCR-SSP (sequence-specific primers) :
Principe :
La réaction de polymérisation en chaîne (PCR) permet une amplification exponentielle in vitro d’une séquence cible de l’ADN à partir de deux amorces oligonucléotidiques complémentaires des extrémités 3’ des deux brins d’ADN encadrant la séquence à amplifier sous l’action d’une ADN polymérase thermostable, chaque amorce est allongée dans le sens 5’->3’.
Chaque cycle d’amplification comporte une succession de trois étapes :
– Dénaturation de l’ADN double brin par la chaleur.
– Hybridation des amorces avec leurs séquences complémentaires. La température d’hybridation est variable selon chaque amorce.
– Extension des amorces par la Taq polymérase qui va incorporer les nucléotides pour synthétiser un nouveau brin d’ADN par le jeu de la complémentarité des bases (A, G, C, T).
La PCR-SSP utilise l’amplification d’un groupe d’allèle avec des amorces spécifiques d’une région polymorphe. La présence ou l’absence d’un allèle est déterminée en fonction de l’amplification ou de la non amplification obtenue.
Cette technique nécessite autant d’amplifications que d’allèles à rechercher.
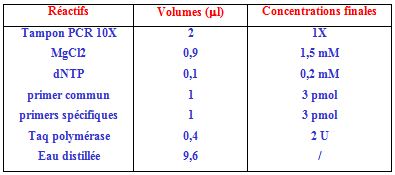
Réactifs :
Pour un volume final de 20 µl, 5 µl d’ADN génomique extrait et dosé à 50 ng/µl, sont ajoutés à 15 µl du mélange réactionnel composé comme suit :
Pour chaque échantillon, on prévoit deux tubes, dans chacun, on ajoute un des deux primers spécifiques et le primer commun.
Primers utilisés :
– Primers spécifiques sens :
IL1αF1 : 5’ CTT TAA TAA TAG TAA CCA GGC AAC AC 3’
IL1αF2 : 5’ CTT TAA TAA TAG TAA CCA GGC AAC AT 3’
– Primer commun anti-sens :
IL1aR: 5’ AAG TAG CCC TCT ACC AAG GA 3’
Programme d’amplification:
Révélation :
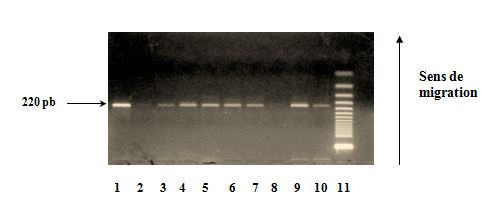
Les produits amplifiés sont révélés par une électrophorèse en gel d’agarose à 2% dans le TBE 1X à 100 volts pendant 45 min et visualisés à l’aide du bromure d’éthidium (BET) sous lumière ultraviolette (UV). La révélation montre une bande spécifique à 220 pb (allèle C ou T) (Fig.3).
Fig.3 : Polymorphisme à la position (-889) du gène de l’IL1-α Vérification de l’amplification sur gel d’agarose à 2%
Puits 1 et 2 : Echantillon n° 1 → C/C.
Puits 3 et 4 : Echantillon n° 2 → C/T.
Puits 5 et 6 : Echantillon n° 3 → C//T.
Puits 7 et 8 : Echantillon n° 4 → C/C.
Puits 9 et 10 : Echantillon n° 5 → C/T.
Puits 11 : Marqueur de poids moléculaire à 50 pb.
b. Polymorphisme à la position +3954 du gène de l’IL1α, du gène de l’Apo 1/Fas et du gène du CTLA-4 par PCR-RFLP (Restriction Fragment Length Polymorphism) :
Principe :
La technique est basée sur :
– Une réaction habituelle de PCR d’une séquence d’ADN cible à partir de deux amorces oligonucléotides complémentaires qui encadrent la séquence cible.
– Une digestion enzymatique du produit amplifié, au niveau des sites de restriction des enzymes spécifiques.
– Une révélation des produits de restriction obtenus.
IL1 β(+3954) :
Réactifs :
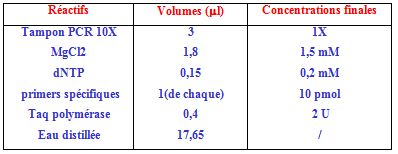
Pour un volume final de 30 µl, 5 µl d’ADN génomique extrait et dosé à 100 ng/µl, sont ajoutés à 25 µl du mélange réactionnel composé comme suit :
Primers utilisés :
IL1 β (1) : 5’ GTT GTC ATC AGA CTT TGA CC 3’
IL1 β (2) : 5’ TTC AGT TCA TAT GGA CCA GA 3’
Programme d’amplification :
Révélation :
Les produits amplifiés sont révélés par une électrophorèse en gel d’agarose à 2% dans le TBE 1X à 100 volts pendant 45 min et visualisés à l’aide du bromure d’éthidium (BET) sous lumière ultraviolette (UV). La révélation montre une bande spécifique à 249 pb (Fig.4).
Fig.4 : Polymorphisme à la position +3954 du gène de l’IL1 β. Vérification de l’amplification sur gel d’agarose à 2%
Tous les échantillons montrent une bande d’amplification à 249 pb sauf l’échantillon n° 11 qui n’est pas amplifié. Puits n° 12 : Marqueur moléculaire à 50 pb.
Digestion enzymatique :
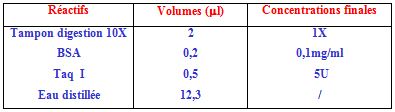
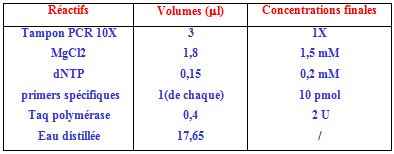
La digestion des produits amplifiés de l’IL1 β (+3954) utilise l’enzyme de restriction Taq I. (Proméga) comme suit : Pour un volume final de 20 µl : 5 µl du produit amplifié, sont ajoutés à 15 µl du mélange réactionnel composé comme suit :
La digestion avec l’enzyme des produits amplifiés se fait à 65°c pendant 5 h.
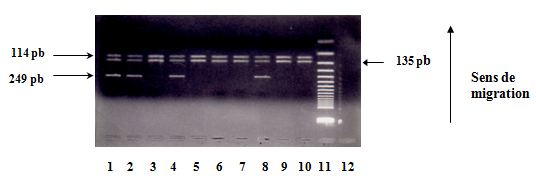
Les produits digérés sont révélés par électrophorèse en gel d’agarose à 4%. La lecture du gel aux UV montre : un fragment non digéré à 249pb (allèle T) et un fragment digéré en deux bandes à 114 et 135 pb (allèle C) (Fig.5).
Fig.5 : Polymorphisme à la position +3954 du gène de l’IL1 β. Révélation des produits de digestion sur gel d’agarose à 4%
Echantillon n° 1, 2, 4 et 8 → Hétérozygote C/T.
Echantillon n° 3, 5, 6, 7, 9 et 10 → Homozygote C/C.
Puits n° 11 → Marqueur moléculaire à 50 pb.
– Apo 1/Fas :
Réactifs :
Pour un volume final de 30 µl, 5 µl d’ADN génomique extrait et dosé à 100 ng/µl, sont ajoutés à 25 µl du mélange réactionnel composé comme suit :
Primers utilisés :
Fas 1 (1) : 5’ CTA CCT AAG AGC TAT CTA CCG TTC 3’
Fas 2 (2) : 5’ GGC TGT CCA TGT TGT GGC TGC 3’
Programme d’amplification :
Révélation :
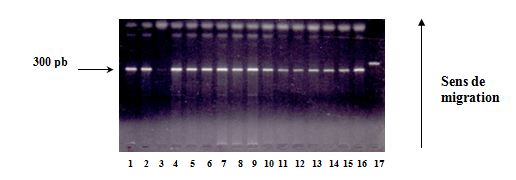
Les produits amplifiés sont révélés par une électrophorèse en gel d’agarose à 2% dans le TBE 1X à 100 volts pendant 45 min et visualisés à l’aide du bromure d’éthidium (BET) sous lumière ultraviolette (UV). La révélation montre une bande spécifique à 300 pb (Fig.6).
Fig.6 : Polymorphisme du gène de l’Apo 1/Fas. Vérification de l’amplification sur gel d’agarose à 2%
Tous les échantillons montrent une bande d’amplification à 300 pb. Puits n° 17 → gel contrôle à 275 pb.
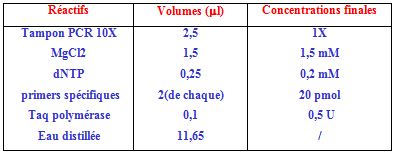
Digestion enzymatique :
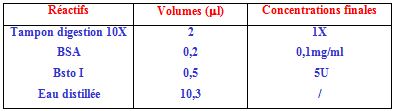
La digestion des produits amplifiés de l’Apo1/Fas utilise l’enzyme de restriction BstoI. (Proméga) comme suit : Pour un volume final de 20 µl : 7 µl du produit amplifié, sont ajoutés à 13 µl du mélange réactionnel composé comme suit :
La digestion avec l’enzyme des produits amplifiés se fait à 60°C pendant 2 h.
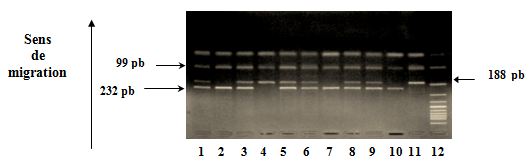
Les produits digérés sont révélés par électrophorèse en gel d’agarose à 3%. La lecture du gel aux UV montre : un fragment de 232 pb (allèle A) et un fragment de 188pb (allèle G) et une bande à 99 pb pour chaque échantillon (Fig.7).
Fig.7 : Polymorphisme du gène de l’Apo 1/Fas. Révélation des produits de digestion sur gel d’agarose à 4%.
Echantillons 1, 3, 5, 6, 8 et 9 → Hétérozygote A/G.
Echantillons 2, 7 et 10 → Homozygote A/A.
Echantillon 4 et 11 → Homozygote G/G.
CTLA-4 (+49) :
Réactifs :
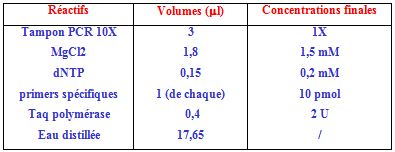
Pour un volume final de 25 µl, 5 µl d’ADN génomique extrait et dosé à 100 ng/µl, sont ajoutés à 20 µl du mélange réactionnel composé comme suit :
Primers utilisés :
CTLA-4 (1) : 5’ CAA GGC TCA GCT GAA CCT GGG T 3’
CTLA-4 (2) : 5’ TAC CTT TAA CTT CTG GCT TTG 3’
Programme d’amplification :
Révélation :
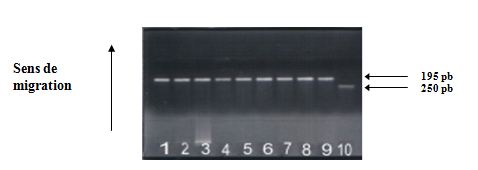
Les produits amplifiés sont révélés par une électrophorèse en gel d’agarose à 2% dans le TBE 1X à 100 volts pendant 45 min et visualisés à l’aide du bromure d’éthidium (BET) sous lumière ultraviolette (UV). La révélation montre une bande spécifique à 195 pb (Fig.8).
Fig.8 : Polymorphisme A/G en position (+49) de l’exon 1 du gène du CTLA-4. Vérification de l’amplification sur gel d’agarose à 2%.
Tous les échantillons montrent une bande d’amplification à 195 pb.
Puits n° 10 → gel contrôle.
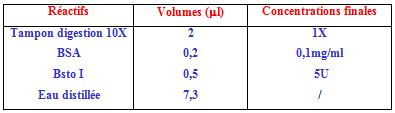
Digestion enzymatique :
La digestion des produits amplifiés du CTLA-4 utilise l’enzyme de restriction kpnI. (Proméga) comme suit :
Pour un volume final de 20 µl : 10 µl du produit amplifié, sont ajoutés à 10 µl du mélange réactionnel composé comme suit :
La digestion avec l’enzyme des produits amplifiés se fait à 37°C pendant 4 h.
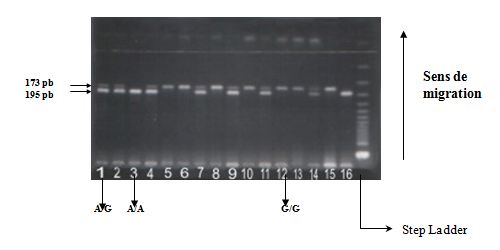
Les produits digérés sont révélés par électrophorèse en gel d’agarose à 3%. La lecture du gel aux UV montre : un fragment de 195 pb (allèle A) et un fragment de 173 pb (allèle G) et une bande à 22 pb pour chaque échantillon (Fig.9).
Fig.9 : Polymorphisme A/G en position (+49) de l’exon 1 du gène du CTLA-4. Révélation des produits de digestion sur gel d’agarose à 4%
(La bande de 22 pb n’est pas toujours observée).
La révélation des produits de digestion se fait sur gel d’agarose 4% dans un tampon TBE (1X) coloré au BET, en présence d’un marqueur de taille (50 pb DNA Step Ladder Promega).
Trois profils électrophorétiques peuvent être observés :
-Une seule bande de 195 pb correspond au génotype homozygote A/A.
-Deux bandes : une de 173 pb et une de 22 pb correspondent au génotype homozygote G/G.
-Trois bandes : une de 195 pb, une de 173 pb et une de 22 pb correspondent au génotype hétérozygote A/G.
c. Polymorphisme du gène du récepteur antagoniste de l’IL1 (IL1Ra) par PCR VNTR (Variable Number Tandem Repeat) :
Principe :
L’intron 2 du gène de l’IL1Ra contient un polymorphisme pentallélique en VNTR, qui consiste en une répétition d’une séquence d’environ 86 pb en tandem. Le nombre de répétitions correspond à un allèle bien spécifique.
Ce polymorphisme a été analysé par PCR simple qui amplifie la séquence répétée en utilisant des amorces spécifiques.
Réactifs :
Pour un volume final de 30 µl, 5 µl d’ADN génomique extrait et dosé à 100 ng/µl, sont ajoutés à 25 µl du mélange réactionnel composé comme suit :
Primers utilisés :
IL1Ra (1) : 5’ TCA GCA ACA CTC CTA T 3’
IL1Ra (2) : 5’ CCT GGT CTG CAG GTA A 3’
Programme d’amplification:
Révélation :
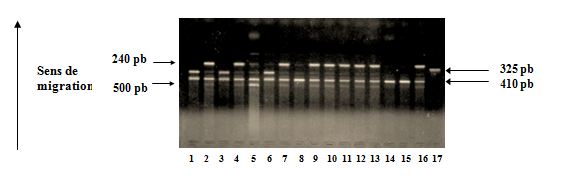
Les produits amplifiés sont révélés par une électrophorèse en gel d’agarose à 2% dans le TBE 1X à 100 volts pendant 45 min et visualisés à l’aide du bromure d’éthidium (BET) sous lumière ultraviolette (UV). La révélation montre une bande spécifique à 410 pb (allèle 1= 4 répétitions de la séquence de 86 pb, une bande à 240 pb (allèle 2= 2 répétitions), une bande à 500 pb (allèle 3= 5 répétions), une bande à 325 pb (allèle 4= 3 répétitions et une bande à 595 pb (allèle 5= 6 répétions) (Fig.10).
A signaler que l’allèle 5 n’a jamais été révélé dans les échantillons testés.
Fig. 10 : Polymorphisme du gène du récepteur antagoniste de l’IL1 (IL1-Ra). Vérification de l’amplification sur gel d’agarose à 2%.
Echantillon n° 1 : 1/4.
Echantillon n° 2 : 1/2.
Echantillon n° 5 : 1/3.
Echantillon n° 8 : 1/1.
Puits n ° 17 : gel contrôle.
VI – Analyses statistiques :
– Fréquences génotypiques : sont obtenues par un comptage direct. On calcule le nombre de sujets présentant un phénotype donné qu’on divise par le nombre total de sujets étudiés.
– Fréquences allèliques : sont le nombre de sujets présentant un allèle donné qu’on divise par le nombre total des allèles dans chaque locus. Chaque individu possède deux allèles dans un locus donné.
– Le test de chi2 (χ²) : est calculé en appliquant la formule :
χ²=∑(Oi-Ci)²/Ci
Avec :
Oi : effectif observé dans chaque cas.
Ci : effectif calculé dans chaque cas.
Si l’un des Ci
χ²=∑((Oi-Ci) – ½)²/Ci
Pour un degré de liberté (ddl) au seuil α= 5%, la valeur de χ² est de 3,84, à partir de laquelle l’association est considérée comme significative et les deux antigènes sont alors en déséquilibre de liaison.